
En esta época, es importante estar a la vanguardia y la secuenciación de nueva generación (NGS) es una plataforma de secuenciación innovadora que puede realizar de cientos a millones de reacciones de secuenciación en paralelo, mientras se analizan simultáneamente los datos de secuenciación resultantes. Recientemente, los Centros de Servicios de Medicare y Medicaid (CMS) han tomado la decisión de cubrir las pruebas de laboratorio de diagnóstico mediante NGS para los pacientes con cáncer avanzado; una decisión que sin duda acelerará la adopción de la NGS en el diagnóstico clínico.
Los científicos empezaron a secuenciar el ADN varios años antes de que los sistemas de gestión de la información de laboratorio (LIMS) estuvieran disponibles comercialmente en 1982 para ayudar a controlar los procesos de laboratorio, el seguimiento de las muestras y los flujos de trabajo y datos del laboratorio. En este artículo hablaremos de cómo el trabajo con el ADN ha avanzado hasta la secuenciación de próxima generación y de cómo las pruebas de laboratorio modernas pueden ayudar a agilizar el proceso de secuenciación del ADN, ofreciendo pruebas de diagnóstico de mayor calidad y un proceso más eficaz.
Hace cuarenta años, la secuenciación del ADN era un proceso manual y muy laborioso que utilizaba materiales radiactivos y sólo generaba secuencias de moléculas de ADN aisladas. Había que conocer de antemano la secuencia de pares de bases de al menos un pequeño tramo del ADN, que se utilizaba como cebador que actuaba como sitio de unión inicial para las enzimas de secuenciación, que generaban la secuencia previamente desconocida. El final de la nueva secuencia se utilizaría como cebador para la siguiente ronda de secuenciación. Con este método, conocido como secuenciación Sanger, se puede secuenciar un gen entero de unos cientos de pares de bases cada vez. El gen medio tiene entre 10 y 15 mil pares de bases, por lo que es fácil apreciar el tiempo que llevaba esto, incluso después de la automatización de la tecnología en 1987.
Las primeras plataformas de secuenciación de nueva generación (NGS), que surgieron a principios de este siglo, se basan en el método Sanger (secuenciación de amplicones), pero son capaces de llevar a cabo de cientos a millones de reacciones de secuenciación en paralelo, junto con el análisis de los datos de secuenciación que se producen. Además de generar cantidades masivas de datos, las plataformas NGS actuales son increíblemente rápidas y relativamente baratas en comparación con las aplicaciones anteriores de secuenciación de ADN. Los costes se reducen continuamente, lo que facilita el uso de la NGS, que es una forma de secuenciación automatizada del ADN, para muchas aplicaciones nuevas que hace sólo 10 años habrían sido prohibitivas desde el punto de vista de los costes. En la actualidad, la NGS se ha trasladado al ámbito sanitario y se utiliza cada vez más para aplicaciones de diagnóstico clínico (por ejemplo, pruebas diagnósticas). Los procesos de NGS de uso común, como la secuenciación del exoma completo, la secuenciación basada en dianas de ADN, la secuenciación del ARN y la secuenciación por inmunoprecipitación de la cromatina, están detrás del desarrollo de procesos y descubrimientos innovadores tanto en el ámbito de la investigación como en el del paciente.
En el ámbito del diagnóstico clínico, la NGS se está explotando en áreas como el diagnóstico de enfermedades hereditarias, la oncología, el asesoramiento genético y la gestión de enfermedades infecciosas. Y fue para una enfermedad hereditaria que las primeras pruebas de NGS fueron aprobadas por la FDA, concretamente, el ensayo MiSeqDx Cystic Fibrosis 139-Variant de Illumina y el ensayo de secuenciación clínica MiSeqDx Cystic Fibrosis. En el ámbito de la oncología clínica, la NGS se utiliza para la elaboración de perfiles tumorales que permiten conocer el pronóstico de la enfermedad y ayudan a tomar decisiones terapéuticas. La NGS también está respaldando el desarrollo de enfoques de medicina personalizada que utilizan ensayos de diagnóstico de acompañamiento basados en la NGS que se emparejan con medicamentos que han sido desarrollados y aprobados para su uso sólo en subconjuntos de pacientes con tumores específicos definidos genéticamente. En consecuencia, este tipo de tecnología también puede ayudar a definir mejor la regulación de los genes.
El primer diagnóstico complementario de NGS que obtuvo la aprobación de la FDA fue el CDxBRCA de Foundation Focus, que ayuda a identificar a las pacientes con tumores de ovario con mutación BRCA para las que se está considerando el tratamiento con Rubraca (rucaparib) de Clovis Oncology. Los dos siguientes diagnósticos complementarios de NGS aprobados por la FDA fueron el Praxis Extended RAS Panel de Illumina, para identificar a los pacientes con cáncer colorrectal que pueden recibir tratamiento con Vectibix, y el Oncomine Dx Target Test de Thermo Fisher Scientific, para seleccionar a los pacientes con cáncer de pulmón no microcítico (CPNM) con mutaciones genéticas específicas.
Las pruebas aprobadas por la FDA tienen un protocolo específico, desarrollado y validado, que hay que seguir. Pero incluso el protocolo de ensayo NGS más sencillo aprobado por la FDA implica un flujo de trabajo complejo con múltiples pasos y procedimientos. Un flujo de trabajo básico de NGS puede seguir un camino a través de múltiples laboratorios que llevan a cabo diferentes funciones, incluyendo la preparación de la muestra, la preparación de la biblioteca de ADN y la secuenciación de amplicones. En este flujo de trabajo de múltiples instalaciones, las muestras se reciben y se registran, y los datos resultantes deben analizarse antes de la publicación de los resultados.
El empleo de un sistema LIMS para gestionar estos flujos de trabajo puede beneficiar en gran medida la eficacia y la eficiencia del laboratorio.
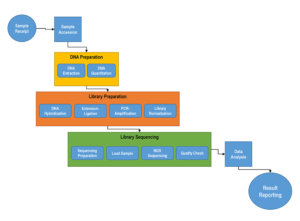
Para los laboratorios que llevan a cabo pruebas NGS y desarrollo de ensayos, la configuración y el ajuste de flujos de trabajo complejos puede llevar mucho tiempo y ser complicado, por lo que tener flujos de trabajo preexistentes que sigan algunos de los procedimientos más estándar es ventajoso. Se introduce una mayor complejidad cuando el laboratorio realiza pruebas NGS para aplicaciones clínicas de secuenciación de ADN. En muchos casos, las pruebas aprobadas por la FDA vienen con características de rendimiento, normas y controles establecidos para demostrar la validez de los resultados. Con algunas pruebas aprobadas por la FDA, como el ensayo MiSeqDx Cystic Fibrosis 139-Variant de Illumina, sigue siendo necesario ver e interpretar los resultados junto con la información adicional del laboratorio y la clínica. Con el desarrollo exponencial y la publicación de ensayos de NGS con fines clínicos, se han desarrollado directrices específicas en torno a los procedimientos asociados a las pruebas de NGS y la notificación de los resultados.
En Estados Unidos, los laboratorios clínicos están sujetos a la supervisión de varios organismos de gobierno. Los Centros de Servicios de Medicare y Medicaid regulan las pruebas de laboratorio a través de las Enmiendas para la Mejora de los Laboratorios Clínicos (CLIA). Además, existen requisitos a nivel estatal que pueden ser más estrictos que los establecidos por la CLIA. También hay organizaciones profesionales de laboratorios, como el Colegio de Patólogos Americanos (CAP), que establece directrices de buenas prácticas para los laboratorios clínicos. Y a medida que la tecnología molecular ha ido avanzando, organizaciones como el Colegio Americano de Genética Médica y Genómica (ACMG) y la Asociación de Patología Molecular (AMP) se unieron al campo.
En 2015, el ACMG y la AMP publicaron una guía conjunta sobre la interpretación de las pruebas genéticas para el diagnóstico clínico¹. Estas directrices recomendaban encarecidamente que las pruebas genéticas moleculares clínicas se realizaran en laboratorios con certificación CLIA e incluían detalles sobre quién puede interpretar los resultados¹. Estas directrices también establecían la necesidad de llevar a cabo la confirmación de los resultados, cuando las variantes de la secuencia se consideran patógenas o probablemente patógenas, tal y como se describe en las directrices¹. Esta publicación hacía referencia a un conjunto anterior de directrices publicadas por el ACMG (2013) específicamente para la NGS (secuenciación automatizada de ADN) que recomendaba métodos para las pruebas de confirmación y proporcionaba orientación para el desarrollo, las metodologías de prueba y validación, y las normas de notificación².
Independientemente del ACMG, en 2014 el CAP añadió 18 nuevos requisitos de la lista de verificación de acreditación de laboratorios a la lista de verificación de patología molecular del CAP³. Los requisitos se aplican a los ensayos basados en NGS en múltiples áreas de enfermedades, incluidos los trastornos hereditarios, la oncología molecular y las enfermedades infecciosas3. Estos requisitos se dividen entre laboratorio húmedo y laboratorio seco y abordan muchos temas relacionados con la NGS, incluidas las pruebas de diagnóstico de confirmación³.
Hay sistemas LIMS que están empezando a acomodar la complejidad de los flujos de trabajo de NGS que implican el procesamiento de muestras multidireccional y múltiples disciplinas dentro del laboratorio. Pero no están necesariamente configurados para gestionar los flujos de trabajo de principio a fin. Para conseguir el resultado final, la NGS depende en gran medida del análisis de datos, que depende de la evaluación inicial del software de secuenciación de amplicones. El análisis inicial requiere determinar las secuencias exactas de las copias individuales de las cadenas de nucleótidos, alinear las lecturas cortas en lecturas más largas superpuestas y, a continuación, comparar las lecturas individuales para identificar las variantes, teniendo en cuenta la intensidad de la señal, los errores y otras métricas de calidad. Se espera que los laboratorios clínicos de NGS establezcan umbrales para las variantes identificadas y la interpretación de si estas variantes son clínicamente significativas o benignas². También se espera que se mantengan altos estándares de calidad para las pruebas de diagnóstico desarrolladas por el laboratorio, y más específicamente la garantía de bajas tasas de falsos positivos.
Según los estándares de laboratorio clínico del ACMG para la secuenciación de nueva generación, “se recomienda que todas las pruebas diagnósticas y/o centradas en la enfermedad incluyan la confirmación del resultado final mediante una tecnología complementaria”². Se espera que, en el futuro, sólo los laboratorios de NGS más experimentados que utilicen algoritmos bien establecidos y probados puedan considerar la eliminación de las pruebas de confirmación con tecnología ortogonal. Por lo tanto, en lugar de llevar a cabo la extensa validación requerida para cada tipo de variante que pueda ser llamada usando NGS, los laboratorios recurren a la secuenciación Sanger como una de las tecnologías más comúnmente empleadas para la confirmación de las pruebas de ADN de la línea germinal. La secuenciación de Sanger es especialmente útil dado que “actualmente se recomienda que todas las pruebas centradas en la enfermedad de los genes de alto rendimiento incluyan una cobertura completa en cada paciente analizado “² , como se encontraría en los paneles de genes y otras pruebas de enriquecimiento de objetivos. Mientras tanto, otros ensayos pueden ser validados mediante pruebas de réplica, a menudo vistas en el análisis de tejidos tumorales o variantes de mosaico, o utilizando tecnologías complementarias como la hibridación in situ fluorescente (FISH).
Muchos laboratorios que han implantado un LIMS para gestionar sus flujos de trabajo de NGS lo han hecho hasta el registro de los resultados de las llamadas de variantes. El objetivo final, tanto si utilizan un LIMS como si combinan varios sistemas, es utilizar las tecnologías informáticas para gestionar todo el proceso de NGS. Desde la extracción de muestras, la creación de bibliotecas y la amplificación, hasta la secuenciación automatizada del ADN, los laboratorios también están empezando a integrar el análisis de datos en sus flujos de trabajo. Sin embargo, las pruebas de confirmación no suelen incluirse en esos flujos de trabajo. Más bien, las pruebas de confirmación suelen tratarse como ensayos separados y puede que ni siquiera se incluyan en los procesos del LIMS que gestiona el flujo de trabajo completo antes de las pruebas de confirmación. Sin embargo, el ACMG recomienda que las pruebas de confirmación se planifiquen con antelación a la necesidad² , mientras que la lista de comprobación del CAP establece que un laboratorio debe contar con políticas para llevar a cabo las pruebas de confirmación necesarias³.
Además, al proporcionar los plazos de entrega de los ensayos previstos, los laboratorios deben tener en cuenta el tiempo necesario para llevar a cabo las pruebas de confirmación. Y en los casos en que los resultados preliminares se publican antes de la finalización de las pruebas de confirmación, se recomienda que el informe indique que las pruebas de confirmación aún no se han realizado. Por ejemplo, “los siguientes resultados no han sido confirmados por un método alternativo o una prueba de repetición”¹.
Para dar cabida a las pruebas de confirmación dentro de un LIMS, pueden establecerse reglas para la siguiente prueba o la prueba de reflejo basadas en los resultados que se devuelven. Lo ideal es que esto forme parte de un flujo de trabajo sin fisuras que no requiera el reenvío de nuevas muestras al laboratorio ni la supervisión manual de la distribución de la metodología. En su lugar, debería seguir las reglas establecidas, redirigir automáticamente las pruebas a la metodología confirmatoria y esperar el resultado confirmatorio antes de la publicación del informe final.
| Por ejemplo |
| Siguiendo las directrices del CAP/ACMG, un panel de 20 genes requeriría que las mutaciones detectadas y las nuevas variantes que superen los requisitos de calidad se confirmen mediante secuenciación Sanger. En esta situación hipotética, la tasa supuesta de pacientes con mutaciones detectadas y llamadas de variantes nuevas es del 10%, y de los 20 genes del panel, las pruebas de confirmación se encuentran en 5 protocolos posibles. Por lo tanto, un laboratorio de alto rendimiento que esté procesando cientos de muestras de pacientes al día podría requerir que el 10% de esas muestras se sometan a algún tipo de prueba de confirmación. En otras palabras, de 500 muestras de pacientes, 50 requerirían la realización de una de las 20 pruebas de confirmación posibles, siguiendo uno de los 5 métodos posibles. |
Los laboratorios de alto rendimiento que utilizan paneles de cáncer multigénicos para evaluar las muestras de los pacientes se enfrentan a flujos de trabajo especialmente difíciles. La secuenciación Sanger (secuenciación de amplicones), que es el método más comúnmente utilizado para las pruebas de confirmación, permite utilizar diferentes pares de cebadores en la misma plataforma, pero es poco probable que todos los posibles pares de cebadores asociados a un panel de genes NGS puedan ser evaluados juntos. El uso de un LIMS para gestionar el número de muestras que pueden requerir pruebas de confirmación sería mucho más fácil cuando ya existen reglas para dirigir las muestras a las diferentes pruebas, y reconocer qué pruebas pueden realizarse simultáneamente, en lugar de tener que añadir manualmente las pruebas y tratar de vincularlas a las muestras originales después del hecho.
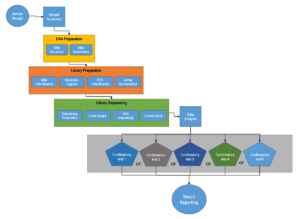
La metodología de la NGS ha permitido el desarrollo de pruebas clínicas avanzadas que pueden informar sobre una atención al paciente más específica y eficaz. Sin embargo, la complejidad de las pruebas basadas en NGS y el análisis de los resultados en un entorno clínico significa que es probable que se requieran pruebas de confirmación. Debería ser una progresión natural para los laboratorios de pruebas clínicas y no clínicas incluir las pruebas de confirmación en sus procesos LIMS. Los flujos de trabajo pueden configurarse a medida que se desarrollan nuevas pruebas y se añaden métodos de validación adicionales.
Aprovechar un LIMS para combinar metodologías de laboratorio de pruebas de muestras basadas en una lógica predefinida sólo puede ayudar a mejorar la eficiencia del laboratorio. Y con la evolución de los métodos de NGS en cuanto a la complejidad de los procesos y los datos, la capacidad de un LIMS para automatizar y gestionar los flujos de trabajo, los procesos y el análisis de los resultados y la elaboración de informes, será aún más valiosa, tanto en los entornos clínicos y de investigación como en las aplicaciones adicionales de la secuenciación del ADN.
Las ventajas son múltiples. La automatización de la selección de métodos de pruebas de confirmación basados en reglas, la programación de las muestras y la elaboración de informes de resultados puede suponer un importante ahorro de costes y tiempo, al tiempo que reduce la introducción manual de datos y la posibilidad de cometer errores. Estos procesos se ven facilitados por la capacidad de secuenciación automática del ADN de la NGS. La automatización del LIMS también reduce la probabilidad de que se pasen por alto accidentalmente las pruebas de confirmación de muestras individuales. El uso de una única plataforma para gestionar todas las muestras y coordinar las pruebas de primera ronda y de confirmación, así como los informes de resultados, ofrece una imagen y un repositorio de datos más completos, a los que se accede a través de un único punto de acceso y que pueden proporcionar informes confidenciales a los médicos y a los clínicos con mayor rapidez.
1. Richards S, Aziz N, Bale S, et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 2015; 17: 405-424.
2. Rehm HL, Bale SJ, Bayrak-Toydemir P, et al. ACMG clinical laboratory standards for next-generation sequencing. Genet Med. 2013; 15: 733-747.
3. Aziz N, Zhao Q, Bry L, et al. College of American Pathologists’ Laboratory Standards for Next-Generation Sequencing Clinical Tests. Arch Path Lab Med. 2015; 139: 481-493.